染色体级基因组揭示了一种入侵性疟疾媒介——斯氏按蚊的隐藏基因组特征
2021年2月10日,BMC Biology在线发表加利福尼亚大学J. J. Emerson实验室文章--斯氏按蚊(Anopheles stephensi)基因组。
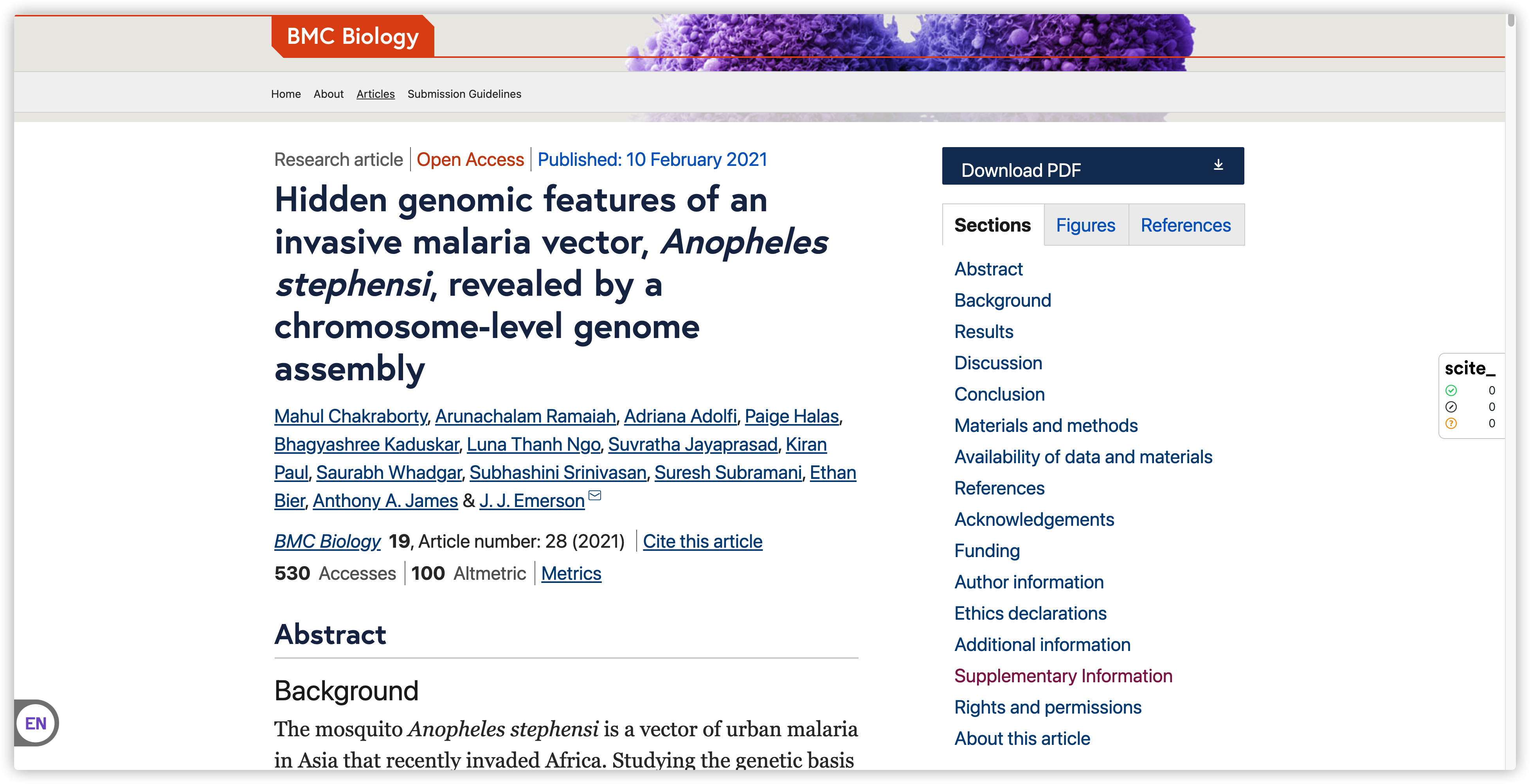
为了获得以前难以处理的基因组区域,我们生成了参考级基因组组装和完整的转录注释,从而达到了疾病媒介昆虫参考基因组的新标准。在这里,我们报道了新的物种特异性转座元件(TE)家族和功能性遗传元件的插入,证明了TEs在基因组进化和表型变异中的广泛作用。我们发现了29个以前隐藏的抗药性基因成员,发现了斯氏按蚊中广泛存在的抗药性的新的候选遗传元件。我们鉴定了2.4Mb的Y染色体和7个新的雄性连锁候选基因,这是所有蚊子中Y染色体覆盖最广的。通过跟踪在血液喂养后超过15天的全长mRNA,我们发现了以前未描述的基因在血液代谢和雌性生殖中的不同作用。Y连锁的异染色质图谱显示,在染色体的进化和退化过程中,长末端重复反转录转座子大量积累。最后,我们确定了一个新的Y连锁的转录因子,它在雄性发育和成年期都有组成性表达,表明它起着重要的作用。
A reference-grade assembly of An. stephensi
- scaffold N50 = 88.7 Mb; contig N50 = 38 Mb
- 99.2% of BUSCOs
- more contiguous and complete
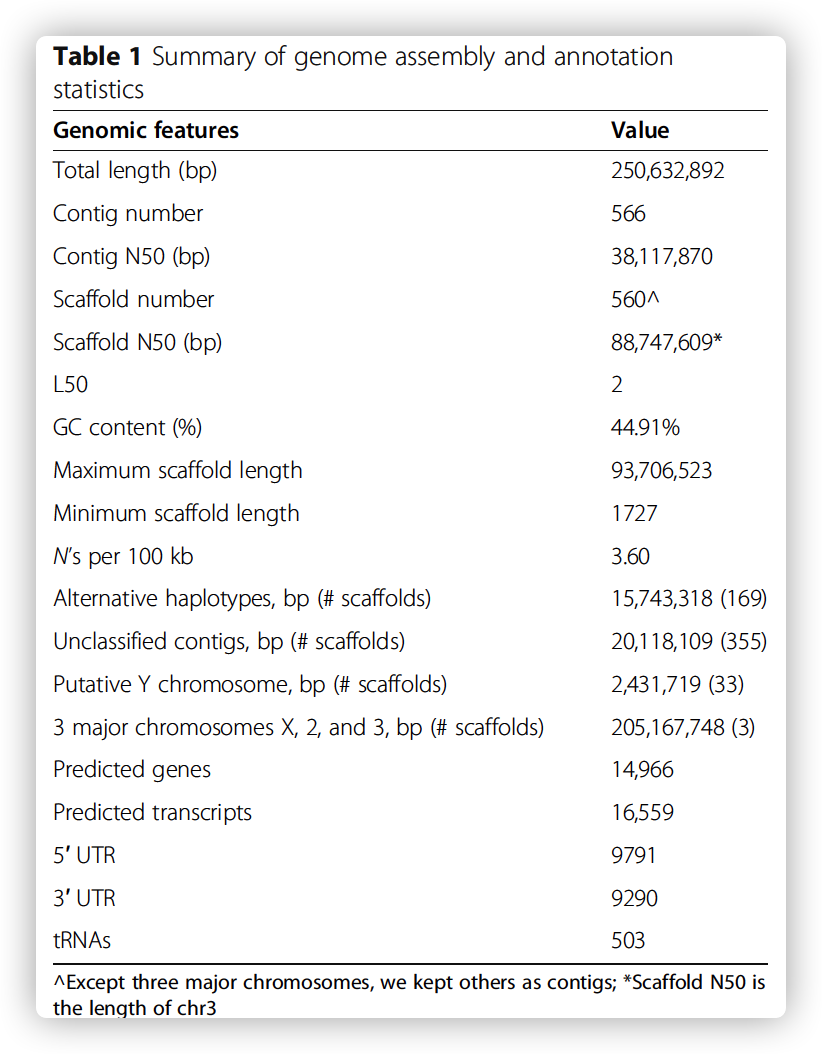
- this assembly comparable to the Drosophila melanogaster reference assembly
- the only Anopheles genome to meet the standards of the Earth BioGenome Project (EBP) for both contiguity and accuracy.
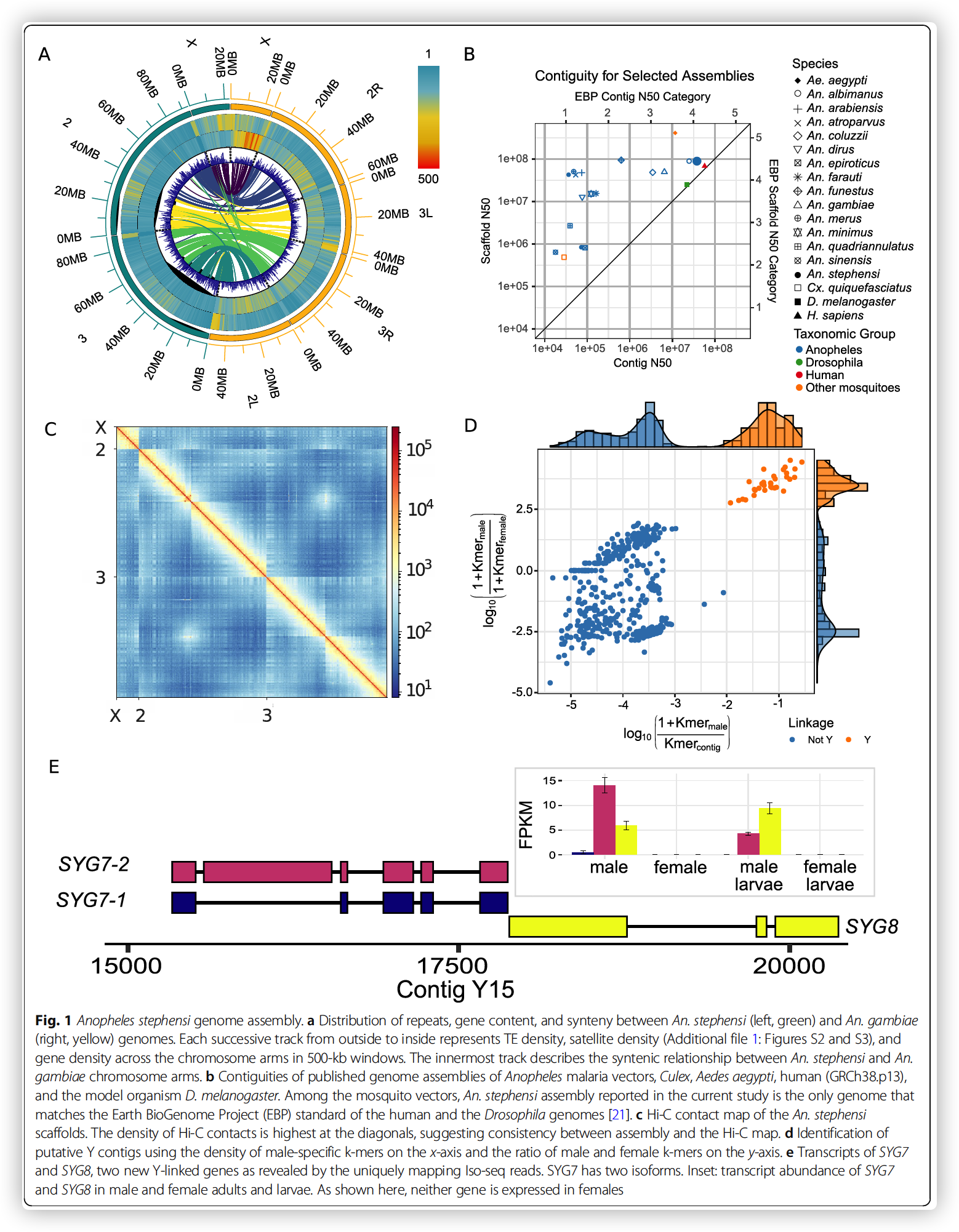
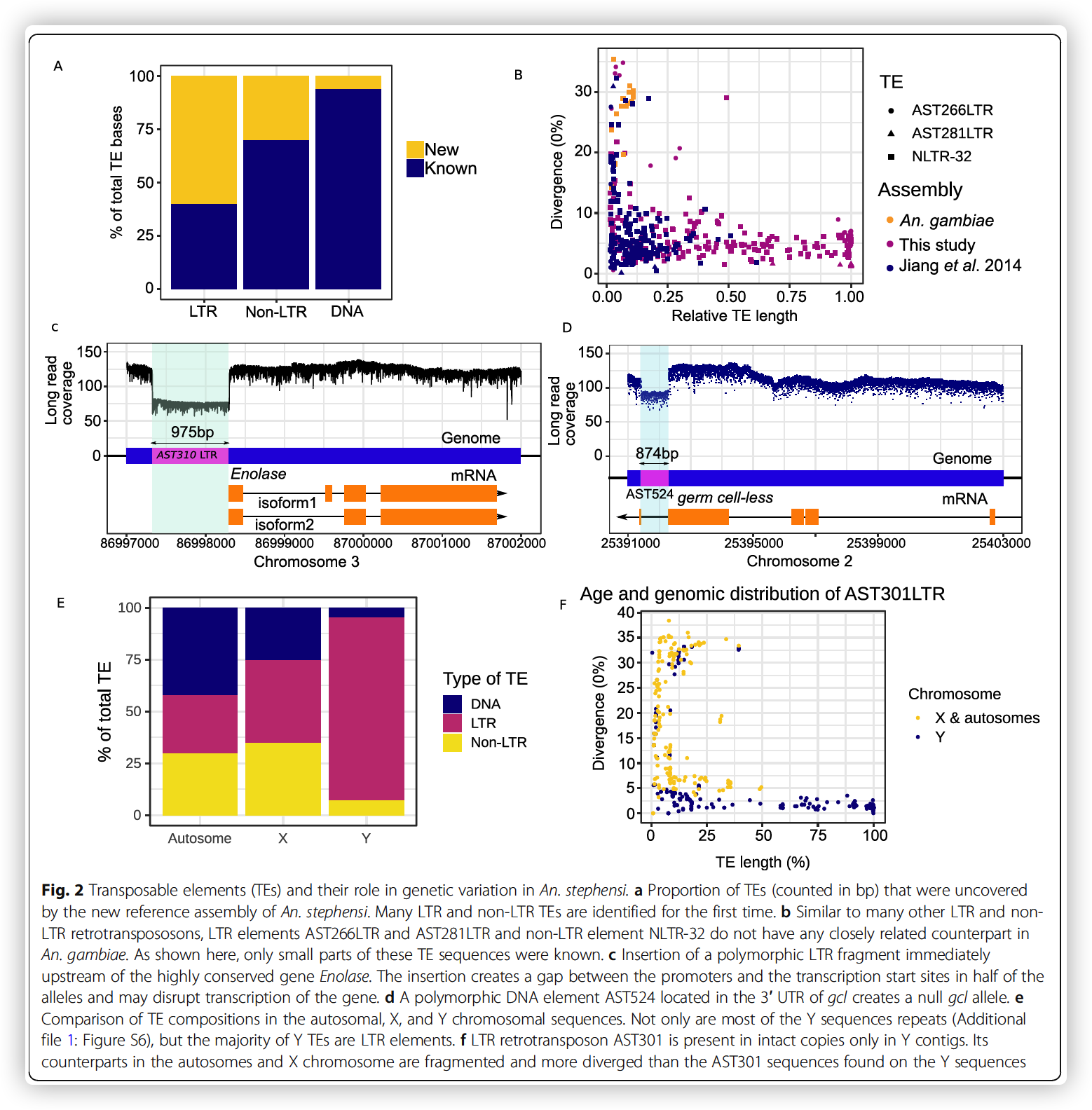
Transposable elements
- Y染色体相关基因可用于蚊子种群的防治
Structural features and newly discovered genes of the Y chromosome
- 利用基于Kmer的方法发现了33个Y contigs
- 72% of the Y sequences (1.7 Mb) comprise LTR elements
- 通过多个独特的Iso-seq序列mapping,发现了7个Y连锁基因
Transcriptional response to blood feeding
另一种旨在减少蚊子数量的抑制方案是修改蚊子种群,以防止它们成为寄生虫媒介。通过喂血在雌性体内诱导的启动子 以被改变用途,以表达阻止疟疾寄生虫传播的效应分子。
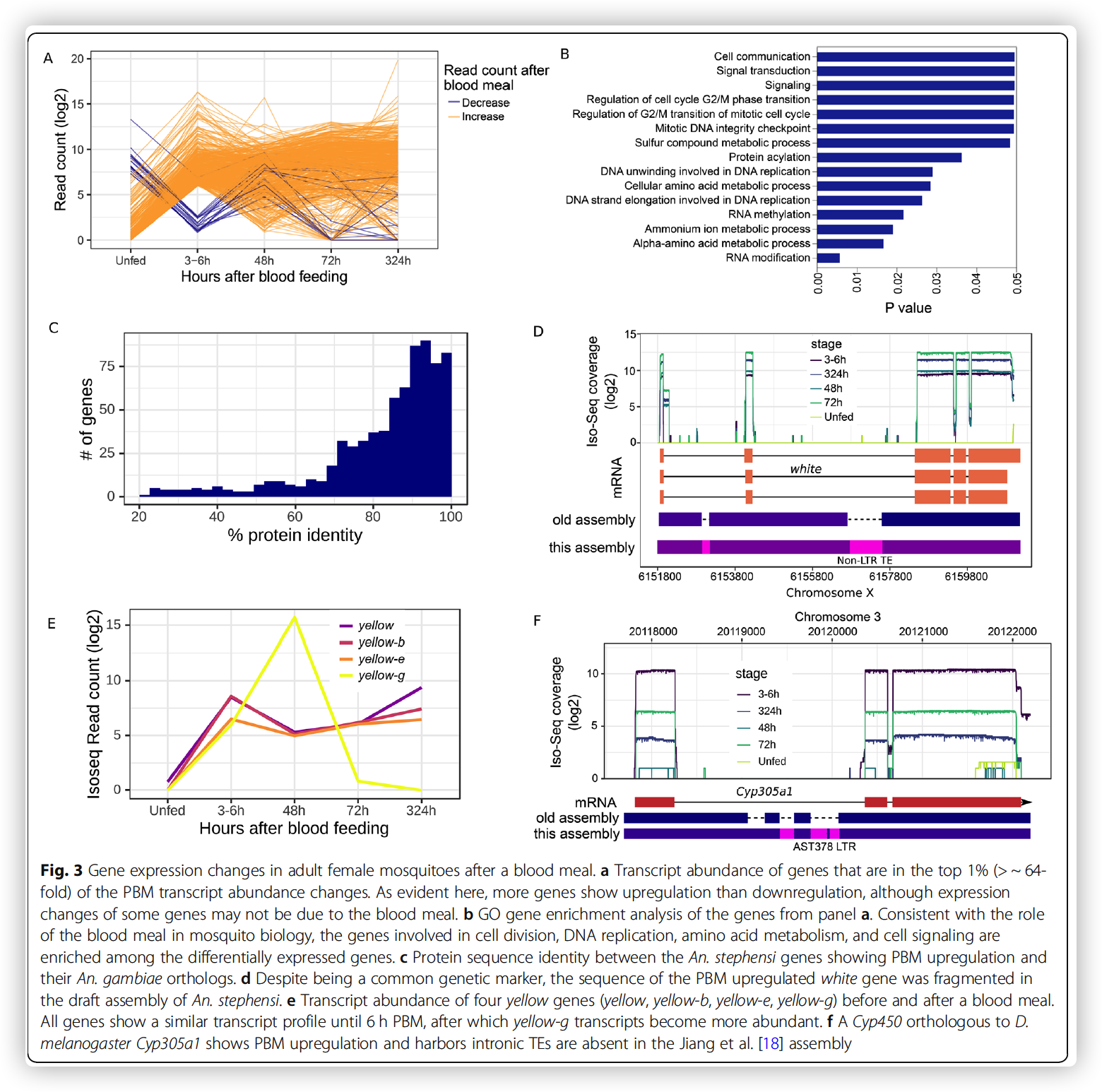
- 吸血后,数百个基因被诱导,其中许多基因在吸血后数天内保持上调
- 593个top 1% 的基因富集到DNA复制,细胞分裂,氨基酸代谢,细胞内和细胞之间的信号转导等
- 上调基因与冈比亚按蚊同源蛋白序列的比较,大多数基因都是相对保守的
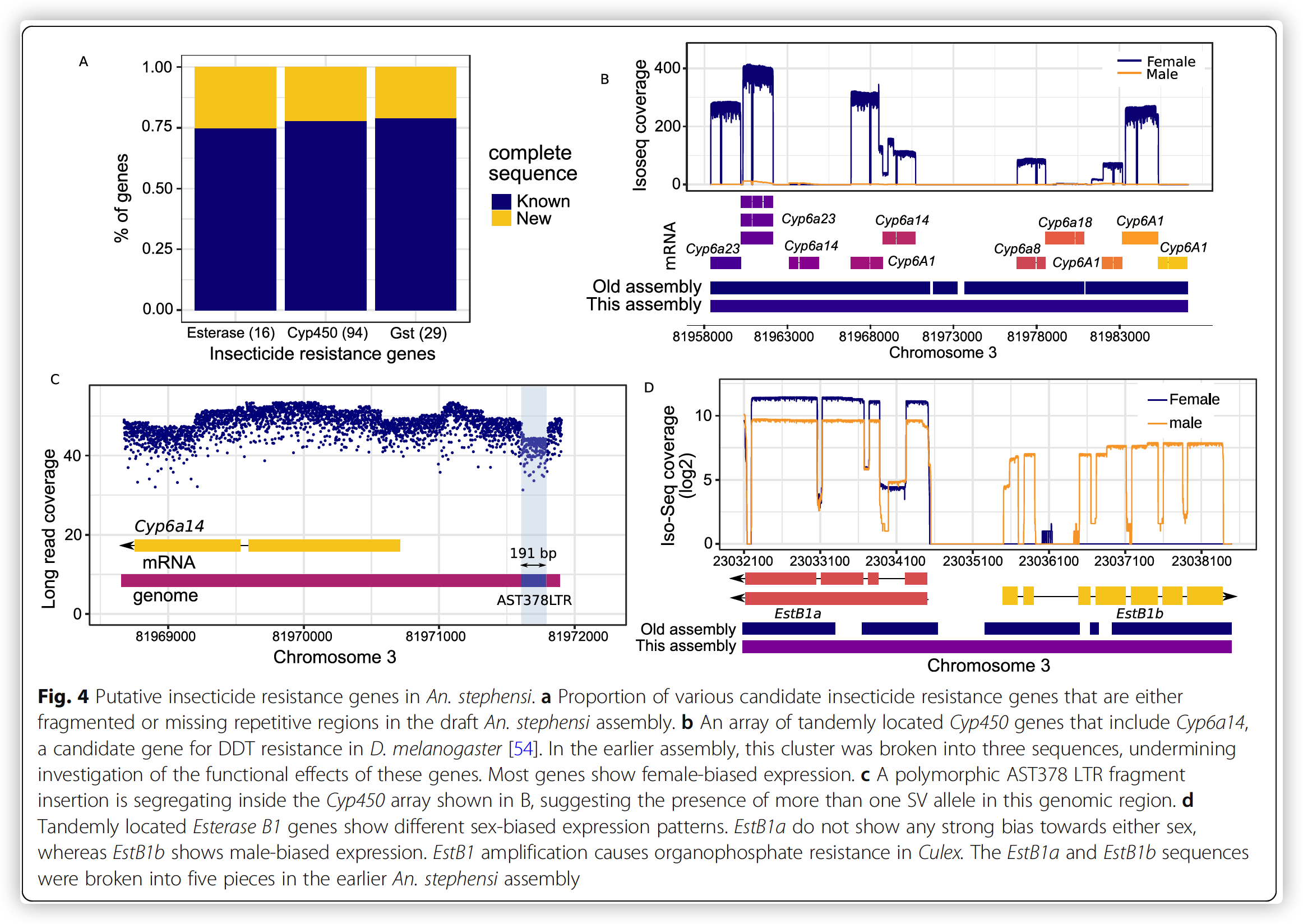
Insecticide resistance genes
- 我们对耐药基因串联序列进行了解析,发现一个由与黑腹果蝇Cyp6a14、Cyp6a23、Cyp6a8、Cyp6a18和家蝇Cyp6A1相似的cyp450组成的28 kb区域
- 一个191-bp的LTR TE多态片段插入转录起始位点5端上游1 kb处,表明在这个复杂区域存在多个SV等位基因
- 我们还解析了先前的酯酶B1片段串联拷贝(Est-B1a和Est-B1b),这些片段已经在致倦库蚊中被证明具有对有机磷的抗性
- 有趣的是,该基因序列中的cyp450和Est-B1b的表达存在性别差异,这表明杀虫剂抗性的分子基础可能存在性别差异
Conclusion
CRISPR和基于基因驱动的策略有望改变对病媒和害虫种群的管理。然而,这些方法的安全性和有效性依赖于对基因组序列及其变体的功能和适合性影响的准确描述。基因组草图不太适合这一目的,因为它们丢失了对这种媒介昆虫的生物学和进化至关重要的重复序列或基因。有关基因正确拷贝或序列的不完整信息可能会误导关于基因或目标序列功能重要性的结论,并可能导致基因驱动中的错误定位或误用。斯氏按蚊基因组揭示了以前未知或未表征的结构和功能基因组元件,这些元件塑造了斯氏按蚊媒介生物学的各个方面。此外,功能上重要的结构变异甚至在这个近交的实验室种群中也是分离的,这表明结构遗传变异在该物种的表型变异中起着重要的作用。最后,最近的技术进步激发了人们对生命之树中所有真核生物测序的热情。它构成了第一个达到这些雄心勃勃的提案所要求的严格参考标准的疟疾媒介,并将与那些针对人和果蝇的既定参考标准并驾齐驱,斯氏按蚊提供了一张全面而准确的基因组功能元件图谱,将为遗传学的新时代奠定基础。
Reference
本文基因组注释的代码如下:https://github.com/mahulchak/stephensi_genome
文章链接:https://doi.org/10.1186/s12915-021-00963-z

